hola no me sale el mismo grafico que el del maestro elimine algunas cosas del código para tenerlo igual como el lo tenia pero no se parece a como el le sale me pueden ayuda profavor
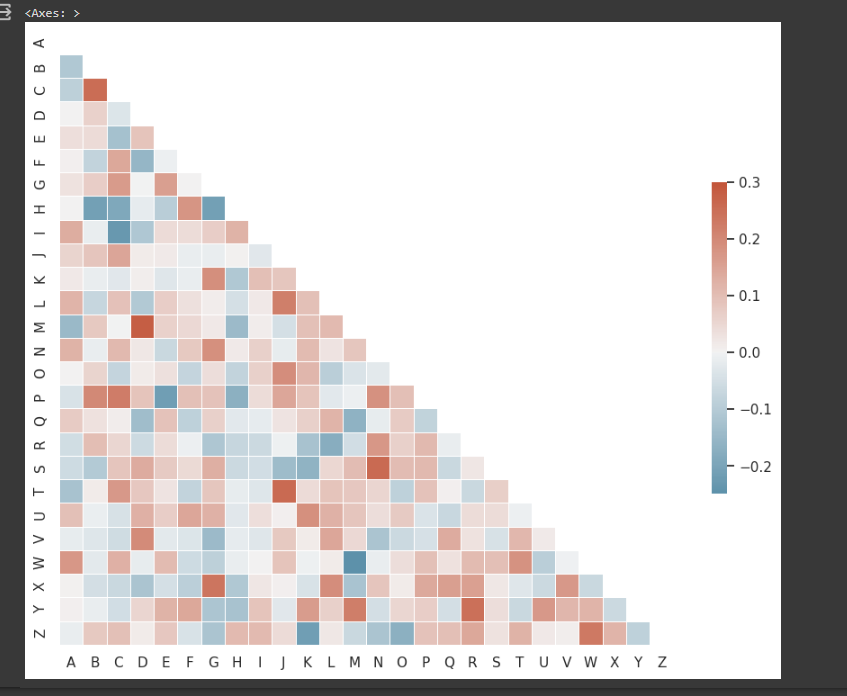
hola no me sale el mismo grafico que el del maestro elimine algunas cosas del código para tenerlo igual como el lo tenia pero no se parece a como el le sale me pueden ayuda profavor


Hola, Isaac!
En ese caso está apareciendo el mismo gráfico del ejemplo de la documentación que el Professor mostró. Para que el gráfico sea igual al que él está presentando, asegúrate de que unos códigos anteriores hayas creado la siguiente variable con el dataset usado en aula:
corr = resultados.corr()
corr
Abrazo y buenos estudios!
Si este post te ayudó, por favor, marca como solucionado ✓.
Hola Erika!
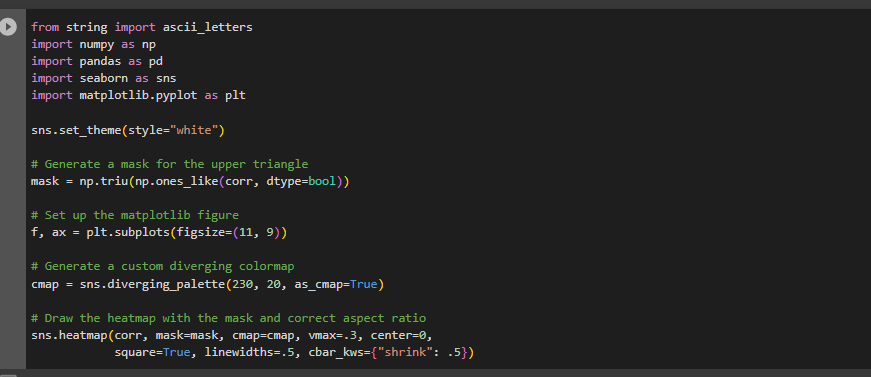
Tengo el mismo problema y ya tenía creada la variable corr = resultados.corr() con anterioridad en el dataset.
Incluso revisé mi código para excluir 2 pasos que se emplean en ejemplo online de Seaborn (https://seaborn.pydata.org/examples/many_pairwise_correlations.html)
# Generate a large random dataset
rs = np.random.RandomState(33)
d = pd.DataFrame(data=rs.normal(size=(100, 26)),
columns=list(ascii_letters[26:]))
# Compute the correlation matrix
corr = d.corr()
Te dejo mi código y agradezco tu ayuda para identificar mi error!
from string import ascii_letters
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
sns.set_theme(style="white")
# Generate a mask for the upper triangle
mask = np.triu(np.ones_like(corr, dtype=bool))
# Set up the matplotlib figure
f, ax = plt.subplots(figsize=(11, 9))
# Generate a custom diverging colormap
cmap = sns.diverging_palette(230, 20, as_cmap=True)
# Draw the heatmap with the mask and correct aspect ratio
sns.heatmap(corr, mask=mask, cmap=cmap, vmax=.3, center=0,
square=True, linewidths=.5, cbar_kws={"shrink": .5})
Tuve la misma situación. Lo solucioné modificando la siguiente línea de código en la función:
modelo = DecisionTreeClassifier(max_depth = max_depth, min_samples_leaf = min_samples_leaf)